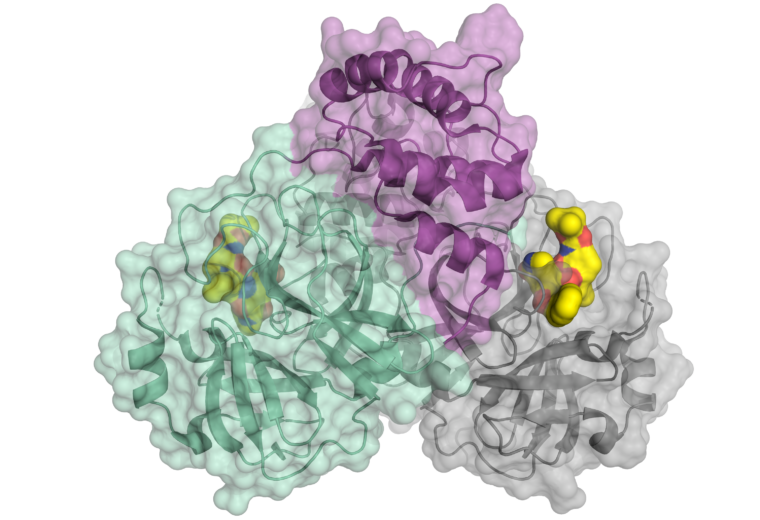
AI löser proteinets gåta
I mer än 50 år har försöken pågått. Nu har forskare med hjälp av AI för första gången lyckats räkna fram strukturen hos okända proteiner.
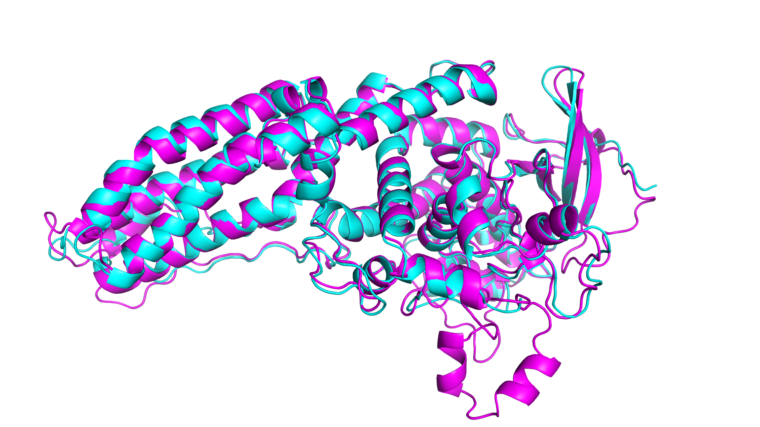
Den lilafärgade strukturen är framräknad med hjälp av Deepminds AI. Den blå är den experimentellt framtagna strukturen.
Bild: CASP/Deepmind/Vincent Tagliabracci and Diana Tomchick, UT South Western
Vartannat år håller organisationen CASP en tävling som går ut på att räkna ut den tredimensionella strukturen hos omkring 100 proteiner. De deltagande lagen får veta i vilken ordning aminosyrorna sitter. Utmaningen är att utifrån denna sekvens räkna ut proteinernas form. Resultaten jämförs sedan med den verkliga strukturen som kartlagts med hjälp av experimentella metoder som röntgenkristallografi och kryoelektronmikroskopi.
Inför årets tävling hade inget lag någon gång tidigare nått samma precision som i laboratoriet. Men när årets resultat presenterades visade det sig att forskarna hos det brittiska och Google-ägda företaget Deepmind för första gången lyckats knäcka problemet som vetenskapen brottats med i mer än 50 år.
Deepminds AI utklassade medtävlarna
Deepminds AI-nätverk utklassade de övriga lagen och kunde förutse strukturen lika bra som experimenten gällande fler än två tredjedelar av proteinerna. Överrensstämmelsen var också bra för den sista tredjedelen, även om den inte nådde samma höga nivå.
Framsteget väntas få stor betydelse inom medicin och bioteknik. Proteiner är livets byggstenar och dess funktion är starkt kopplat till deras tredimensionella struktur.
Av alla proteiner som finns i naturen är strukturen bara känd hos en bråkdel. Att kartlägga ett enda protein är ett mödosamt arbete som kan ta flera år. Med datorns hjälp kan samma uppgift lösas på en halvtimma.
På Deepminds blogg framgår att AI-nätverket, som kallas Alpha Fold, tränades på omkring 170 000 proteiner med känd struktur samt ett stort antal där strukturen inte är känd.
Alpha Fold har tidigare använts för att räkna fram strukturen på flera proteiner hos sars-cov-2-viruset.

Kunskap baserad på vetenskap
Prenumerera på Forskning & Framsteg!
Inlogg på fof.se • Tidning • Arkiv med tidigare nummer