Kemipristagarna har avkodat proteinernas språk
De har löst en av biokemins största gåtor – hur aminosyror veckas till funktionella proteiner. Årets kemipristagare gör det enklare att studera proteiner, men också att designa nya.

David Baker delar kemipriset med Demis Hassabis och John Jumper.
Bild: University of Washington, Getty images
Svaret på biokemins stora utmaning. Så beskriver Heiner Linke, ordförande för Nobelkommittén i kemi, årets Nobelpris i kemi på presskonferensen.
Pristagare är David Baker ”för datorbaserad proteindesign” och Demis Hassabis och John Jumper på Google Deepmind ”för proteinstrukturprediktion” med AI-verktyget Alphafold.
Strukturen avgör proteinets funktion
Proteiner består av strängar av aminosyror. De här strängarna är veckade och skapar en unik tredimensionell struktur för proteinet som avgör dess funktion.
Cyrus Levinthal konstaterade 1968 att ett protein med 100 aminosyror skulle kunna anta 10^47 olika strukturer, beroende på hur strängen veckas. För ett protein med 100 aminosyror skulle det då ta längre tid än hela universums ålder att hitta ”rätt” struktur. I själva verket tar det millisekunder.
Förklaringen blir att det måste finnas något i själva sekvensen av aminosyrorna som bestämmer hur de veckas, men exakt hur är en gåta biokemister har brottats med i flera decennier. Vad är kopplingen mellan koden och funktionen för ett protein?
Med datorhjälp har årets nobelpristagare i kemi äntligen lyckats svara på det här.

Bild: Johan Bodell
– De har i princip löst proteinernas språk. Vi har kunnat bokstäverna, aminosyrorna, sedan tidigare, men vi har inte fattat hur de talar med varandra och leder till de här funktionella formerna, säger Pernilla Wittung-Stafshede, professor i kemi vid Chalmers tekniska högskola och ledamot i Nobelkommittén för kemi.
Att designa nya proteiner
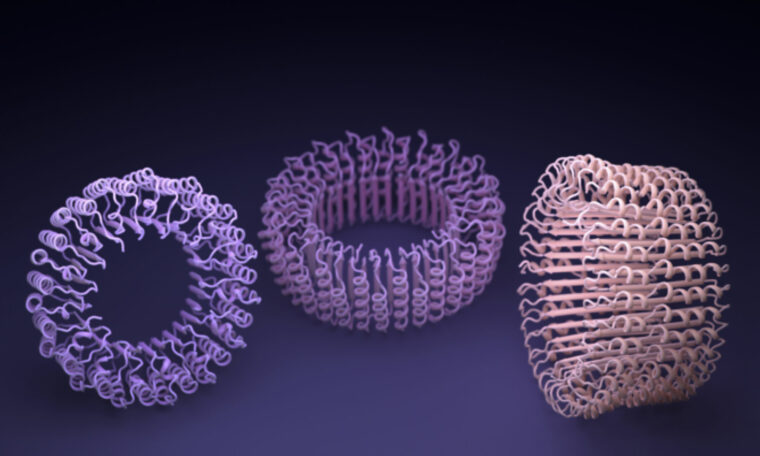
David Bakers forskning kom först. Redan 2003 presenterade hans team ett helt nytt protein: Top7 – det första protein som designats av en människa utan en förlaga i naturen.
Illustration: Terezia Kovalova / Kungl. Vetenskapsakademien
Den föreslagna 3D-strukturen för proteinet matades in i David Bakers egenkonstruerade program Rosetta, som jämförde strukturen med fragment av andra proteinstrukturer i databaser och föreslog hur aminosyrasekvensen borde se ut. Med hjälp av den datagenererade aminosyrasekvensen kunde de senare producera Top7 i bakterier.
Sedan dess har David Bakers team designat flera nya proteiner som använts i allt från vaccin till biosensorer.
– Sen kom den här strukturprediktionen – Alphafold – och det har man fört in i designmetoderna också. Nu kan man göra ännu bättre designproteiner, säger Pernilla Wittung-Stafshede.
Från sekvens till funktion
Strukturprediktionen – som Demis Hassabis och John Jumper belönas för – är andra änden av priset. Den har besannat biokemisternas dröm om att kunna titta på ordningen av aminosyror och därifrån kunna säga hur proteinet ser ut.
2016 började AI-forskarna på Google Deepmind i hemlighet undersöka vad de kunde bidra med. De matade sitt artificiella neuronnät med träningsdata baserat på alla de proteinstrukturer och proteinsekvenser som redan fanns publicerade. AI:n jämförde sedan en given aminosyrasekvens med andra proteiner för att hitta gemensamma nämnare, och kunde utifrån det skapa en karta över hur långt det är mellan varje aminosyra i proteinet. Den här kartan blev sedan grunden till en tredimensionell struktur.
– AI är ju själva genombrottet här. Man kunde träna upp modellen på publicerade aminosyrasekvenser och strukturer och sedan använder man den här AI:n – som kan associera och fokusera och förstå de här mönstren och spotta ur sig en struktur, säger Pernilla Wittung-Stafshede.
Alphafold 2, som lanserades 2021, kunde förutsäga strukturer med lika liten felmarginal som de klassiska teknikerna som röntgenkristallografi och kryoelektronmikroskopi. Skillnaden är bara att de klassiska teknikerna i bästa fall tar veckor, ofta år, för att bestämma ett proteins struktur. Alphafold gör det på minuter. Nästa modell, Alphafold 3, lanserades i maj i år.
När Alphafold 2 lanserades gjordes den också öppen för alla att använda. Det har gjort att den har fått ett enormt genomslag på kort tid. En som dagligen använder tekniken i sin forskning är Ingemar André, professor i biokemi vid Lunds universitet.
– Det finns nog väldigt få forskare inom livsvetenskaperna som håller på med proteiner som inte har använt Alphafold-metodiken för att studera proteiners struktur. Det har gått från en nischmetod – som kan användas av få personer och med osäkra resultat – till en standardmetodik för alla inom biologiska discipliner, säger Ingemar André.
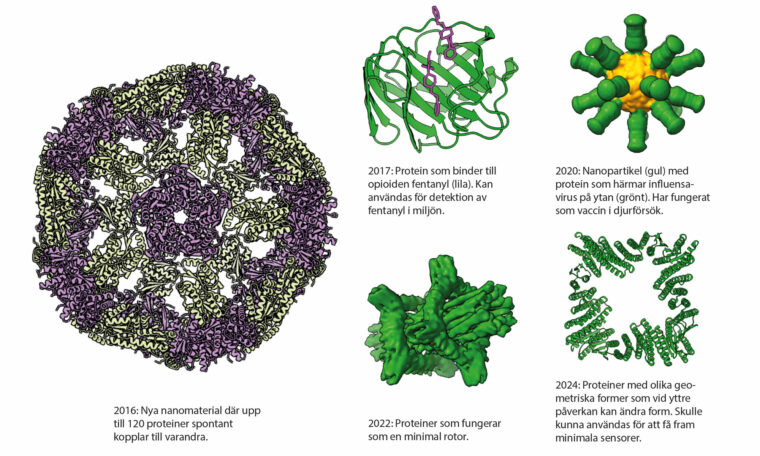
Proteiner och proteinstrukturer framtagna av Rosetta och Alphafold 2

Så fungerar Alphafold 2
Kemipriset 2024: David Baker, Demis Hassabis och John Jumper

”för datorbaserad proteindesign”
David Baker föddes 1962 i Seattle, USA, och doktorerade 1989 vid University of California, Berkeley, USA, och är professor vid University of Washington, Seattle, USA.
”för proteinstrukturprediktion”
Demis Hassabis föddes 1976, i London, Storbritannien och doktorerade 2009 vid University College London, Storbritannien. Han är vd för Google Deepmind i London, Storbritannien.
John M. Jumper föddes 1985 i Little Rock, USA, och doktorerade 2017 vid University of Chicago, USA. Han är senior research scientist på Google Deepmind i London, Storbritannien.
Här nedan kan du se direktsändningen av tillkännagivandet i efterhand.

Kunskap baserad på vetenskap
Prenumerera på Forskning & Framsteg!
Inlogg på fof.se • Tidning • Arkiv med tidigare nummer